réservé à la recherche
Cl-amidine inhibiteur de PAD
N° Cat.S8141
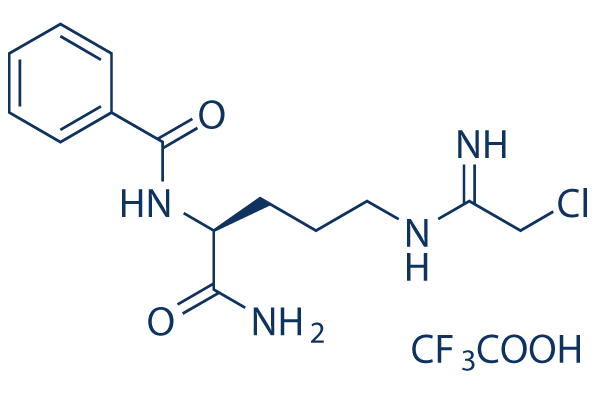
Structure chimique
Poids moléculaire: 424.8
Contrôle qualité
| Cibles apparentées | PD-1/PD-L1 CXCR STING AhR CD markers Interleukins Anti-infection Antioxidant COX Histamine Receptor |
|---|---|
| Autre Immunology & Inflammation related Inhibiteurs | Bestatin (Ubenimex) Bindarit (AF 2838) Tranilast Tempol Sinomenine GI254023X (GI4023) ATP Geniposidic acid CORM-3 Oxymatrine |
Culture cellulaire, traitement et concentration de travail
| Lignées cellulaires | Type dessai | Concentration | Temps dincubation | Formulation | Description de lactivité | PMID |
|---|---|---|---|---|---|---|
| MDA-MB-231 | Cytotoxicity assay | 400 uM | 96 hrs | Cytotoxicity against human MDA-MB-231 cells assessed as decrease in cell number at 400 uM after 96 hrs by trypan blue assay | 23420624 | |
| MDA-MB-231 | Cytotoxicity assay | 200 to 400 uM | 96 hrs | Cytotoxicity against human MDA-MB-231 cells assessed as decrease in cell viability at 200 to 400 uM after 96 hrs by trypan blue assay | 23420624 | |
| HL60 | Function assay | 5 to 10 uM | 15 mins | Inhibition of PAD4 in human HL60 cells assessed as reduction in A23187-induced citrullinated H4 level at 5 to 10 uM preincubated for 15 mins followed by A23187 addition measured after 15 mins by Western blot analysis | 30344909 | |
| HEK293T | Function assay | 10 nM to 100 uM | Inhibition of human recombinant PAD3 expressed in HEK293T cells assessed as conversion of BAEE to sodium benzoyl-L-citrulline at 10 nM to 100 uM by colorimetric analysis | ChEMBL | ||
| HEK293T | Function assay | 100 uM | 24 hrs | Inhibition of thapsigargin-induced cell death in human HEK293T cells overexpressing human recombinant PAD3 assessed as increase in cell survival at 100 uM treated 15 mins prior to thapsigargin addition measured after 24 hrs drug treatment by methylene blu | ChEMBL | |
| Cliquez pour voir plus de données expérimentales sur les lignées cellulaires | ||||||
Informations chimiques, stockage et stabilité
| Poids moléculaire | 424.8 | Formule | C14H19ClN4O2.C2HF3O2 |
Stockage (À partir de la date de réception) | |
|---|---|---|---|---|---|
| N° CAS | 1043444-18-3 | -- | Stockage des solutions mères |
|
|
| Synonymes | N/A | Smiles | C1=CC=C(C=C1)C(=O)NC(CCCN=C(CCl)N)C(=O)N.C(=O)(C(F)(F)F)O | ||
Solubilité
|
In vitro |
DMSO
: 84 mg/mL
(197.74 mM)
Ethanol : 84 mg/mL Water : 35 mg/mL (Ultrasonic and heating.) |
Calculateur de molarité
|
In vivo |
|||||
Calculateur de formulation in vivo (Solution claire)
Étape 1 : Entrez les informations ci-dessous (Recommandé : Un animal supplémentaire pour tenir compte des pertes pendant lexpérience)
Étape 2 : Entrez la formulation in vivo (Ceci nest que le calculateur, pas la formulation. Veuillez nous contacter dabord sil ny a pas de formulation in vivo dans la section Solubilité.)
Résultats du calcul :
Concentration de travail : mg/ml;
Méthode de préparation du liquide maître DMSO : mg médicament prédissous dans μL DMSO ( Concentration du liquide maître mg/mL, Veuillez nous contacter dabord si la concentration dépasse la solubilité du DMSO du lot de médicament. )
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuiteμL PEG300, mélanger et clarifier, ajouter ensuiteμL Tween 80, mélanger et clarifier, ajouter ensuite μL ddH2O, mélanger et clarifier.
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuite μL Huile de maïs, mélanger et clarifier.
Remarque : 1. Assurez-vous que le liquide est clair avant dajouter le solvant suivant.
2. Assurez-vous dajouter le(s) solvant(s) dans lordre. Vous devez vous assurer que la solution obtenue lors de lajout précédent est une solution claire avant de procéder à lajout du solvant suivant. Des méthodes physiques telles que le vortex, les ultrasons ou le bain-marie peuvent être utilisées pour faciliter la dissolution.
Mécanisme daction
| Targets/IC50/Ki |
PAD1
(Cell-free assay) 0.8 μM
PAD4
(Cell-free assay) 5.9 μM
PAD3
(Cell-free assay) 6.2 μM
|
|---|---|
| In vitro |
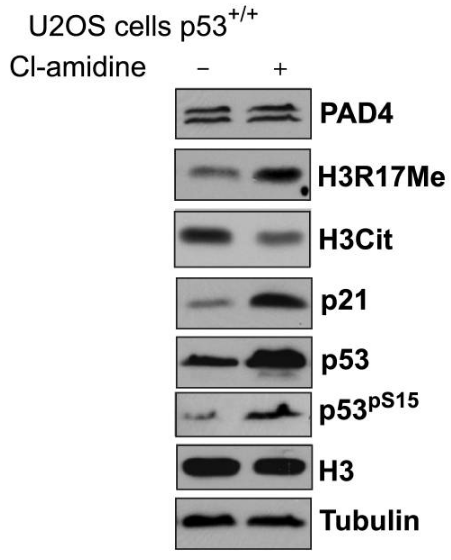
La Cl-amidine antagonise l'amélioration de l'interaction p300GBD-GRIP1 médiée par PAD4 de manière dose-dépendante. L'effet inhibiteur de ce composé n'est pas non spécifique mais cible l'enzyme PAD4 active. Ce composé augmente l'expression de p53 dans les cellules immunitaires CD45 positives. Il déclenche la différenciation et l'apoptose de multiples lignées cellulaires cancéreuses p53+/+ et p53−/− (par exemple, les cellules HL60, HT29, TK6 et U2-OS). Ce produit chimique induit l'expression de p53 et de plusieurs gènes cibles en aval, notamment l'inhibiteur de kinase dépendant de la cycline p21, GADD45, et la protéine pro-apoptotique PUMA dans les cellules d'ostéosarcome U2-OS.
|
| In vivo |
Le traitement par Cl-amidine inhibe la formation de NET (piège extracellulaire de neutrophiles) de NZM (New Zealand mixed 2328) in vivo et modifie significativement les profils d'auto-anticorps circulants et les niveaux de complément tout en réduisant le dépôt d'IgG glomérulaires. De plus, ce composé augmente la capacité de différenciation des cellules progénitrices endothéliales de la moelle osseuse, améliore la vasorelaxation dépendante de l'endothélium et retarde considérablement le temps jusqu'à la thrombose artérielle induite par une lésion photochimique. Il retarde le développement de la thrombose chez les souris NZM. Ce produit chimique inhibe les PAD chez les souris sans toxicité significative et améliore les phénotypes de la maladie dans des modèles animaux d'arthrite inflammatoire et de maladie inflammatoire de l'intestin. Et il a été démontré qu'il réduit la gravité de la maladie dans des modèles murins de colite ulcéreuse et de PR.
|
Références |
|
Applications
| Méthodes | Biomarqueurs | Images | PMID |
|---|---|---|---|
| Western blot | PAD4 / H3R17Me / H3Cit / p21 / p53 / p-p53 iNOS MMP-2 / MMP-9 p-Elk-1 / Elk-1 |
|
18505818 |
Support technique
Tel: +1-832-582-8158 Ext:3
Si vous avez dautres questions, veuillez laisser un message.
